La resistenza agli antibiotici è una minaccia reale per la salute pubblica e, in assenza di strategie mirate, avrà un impatto ancora maggiore dell'attuale pandemia.
Per i non addetti ai lavori tale affermazione potrebbe sembrare una esagerazione ma i numeri parlano chiaro. Negli ultimi decenni la diffusione dei cosiddetti superbatteri (alcuni dei quali resistenti a tutti gli antibiotici noti) provoca ogni anno (e parlo del solo mondo sviluppato, decine di migliaia di morti riportando le lancette dell'orologio per alcune malattie a prima degli anni '40 quando una qualunque ferita poteva degenerare in infezione fatale.
Numeri esplicativi. I superbatteri sono la quarta causa di morte negli USA e ci si riferisce alle sole infezioni "classiche" (TBC et similia escluse) contratte spesso e volentieri durante il ricovero in ospedale. Uno studio dell'università di Washington del 2018 ha quantificato in 162 mila i decessi correlati, un numero più del doppio di quello causato dall'abuso di oppioidi e farmaci correlati.
I batteri responsabili non sono nulla di esotico ma comuni come lo Staphylococcus aureus o l'Enterococcus faecium la cui infezione era fino a pochi anni fa facilmente gestibile con un singolo trattamento. A questi andrebbero poi aggiunti i batteri (per ora) meno diffusi in occidente come quello della tubercolosi, di cui tuttavia sono noti ceppi resistenti a 7 antibiotici diversi, fenomeno di cui ho scritto nel lontano 2013("Il passato che ritorna: polio e tbc")
Come descritto le cause sono molteplici anche se possiamo identificarne 2 principali (per una trattazione più completa --> "la sfida continua con i microbi"):
- l'utilizzo non giustificato, massiccio e spesso incompleto di antibiotici sia negli allevamenti che nell'uso domestico;
- la ricerca che non ha prodotto nuovi antibiotici in decenni. Un ritardo causato dagli alti costi necessari per un ritorno economico insufficiente a coprire anche solo le spese, deterrente sufficiente a fare scappare le aziende biotech.
Tra i due è il primo a giocare un ruolo chiave in quanto "nursery" di nuove resistenze che da sole vanificherebbero gli sforzi della più efficiente R&D. A questo aggiungiamo la capacità che hanno molte specie di batteri di scambiare tra loro (inteso tra specie non correlate) il materiale genetico responsabile sia della virulenza che della resistenza agli antibiotici; facile capire allora la diffusione a livello globale della resistenza agli antibiotici con la trasformazione in superbatteri di batteri prima "innocui".
In termini tecnici si parla di trasmissione orizzontale oltre alla classica trasmissione verticale.
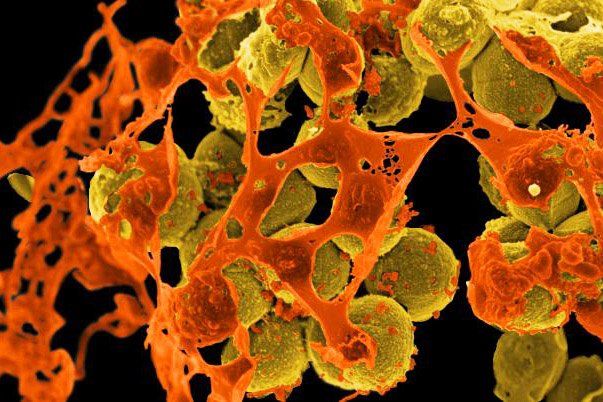 |
| Micrografia elettronica a scansione di Staphylococcus aureus resistente alla meticillina (MRSA) circondato da detriti cellulari. L'MRSA è un tipico esempio di superbatterio (Image credit: NIAID via UCSF) |
Come mai esistono geni per la resistenza?
La maggior parte degli antibiotici (in primis quelli classici) deriva da molecole prodotte da altri batteri (streptomiceti, ...) come arma contro altri tipi di batteri sia come modo di "predazione" che per eliminare la concorrenza dalle risorse ambientali. Ogni batterio sensibile ad un dato antibiotico non ha altra scelta, pena l'estinzione, che "evolvere" delle difese: enzimi in grado di distruggere l'antibiotico (ad esempio la beta-lattamasi); pompe che espellono la molecola dalla cellula oppure modifiche del target dell'antibiotico tale da rendere quest'ultimo inefficace.
Soluzioni
Il primo, e tardivo, passo è stato cambiare i comportamenti che hanno causato la forte pressione selettiva dei superbatteri: divieto di uso antibiotici senza prescrizione; parsimonia negli allevamenti e impianti di bonifica di tutti i reflui (inclusi quelli ospedalieri).
Occorreva però anche un cambio di paradigma nella R&D per rendere il processo economicamente sostenibile e veloce.
Esattamente quello che è stato fatto dai ricercatori della UCSF con un approccio che potremmo definire in stile "LEGO": riprogettare le molecole antibiotiche esistenti per eludere i meccanismi di resistenza di un batterio, partendo da unità "LEGO molecolari" assemblabili per per formare molecole più grandi e funzionali.
La ricerca è stata pubblicata lo scorso settembre sulla rivista Nature.
L'approccio rientra nel solco di strategie note come directed evolution e dynamic combinatorial libraries finalizzate a sfruttare criteri di auto-selezione di molecole sempre più efficaci partendo da librerie di molecole "messe sotto pressione evolutiva" (qui una ottima review). Per una sintesi degli approcci usati in passato per gli anitbiotici vedi History of antimicrobial discovery.
I ricercatori californiani avevano due obiettivi: ridare vita a classi di farmaci il cui potenziale era andato scemando nel tempo per la perdita di efficacia degli antibiotici o il riposizionamento di farmaci (drug repurposing) in uso per altri scopi ma di cui vi erano indizi di attività antimicrobiche.
Il valore aggiunto per alcune di queste molecole è l'essere già state testate in esseri umani, quindi con profilo di sicurezza noto.
Il vantaggio reale di questo approccio è che potrebbe, se i risultati saranno all'altezza delle aspettative, eliminare la necessità di inventare nuove classi di antibiotici agendo direttamente sulla molecola per inficiare la resistenza batterica.
Sia chiaro, questo non è un approccio risolutivo perché i batteri (e i virus ancora di più nel caso degli antivirali e dei vaccini) hanno tempi e numeri di replicazione tali da mettere sotto stress contromisure alle nostre contromisure, a cui ci si troverà a dovere rispondere con altre modifiche. Ma questo è un problema ineludibile, parte intrinseca delle sfide evolutive a cui i batteri, in giro da 3 miliardi di anni e presenti in ogni nicchia biologica, da sempre fanno fronte. Il vero scopo è minimizzare il nostro tempo di reazione e (dove possibile, attraverso trattamenti combinati) la comparsa ex ante della resistenza.
Il gruppo guidato da Ian Seiple ha testato l'approccio (vera e propria proof of concept) su una classe di antibiotici chiamati streptogramine. Scelta non casuale se si pensa che le streptogramine sono state per anni un'arma molto efficace contro le infezioni da S. aureus ... finché i batteri non hanno sviluppato un meccanismo per neutralizzarle.
 |
| La Virginiamycin M1è un membro dell'ampia classe di streptogramine (credit:wikipedia) |
Un breve accenno a come funzionano le streptogramine. Per dirla in termini semplici è come se "incollassero" la parte catalitica del ribosoma batterico, rendendo loro impossibile la produzione di proteine (azione citostatica, citotossica se associata ad altro antibiotico). I ceppi resistenti hanno evoluto come contromisura la produzione di proteine come le virginiamicina acetiltransferasi (Vats), che riconoscono le streptogramine non appena entrano nella cellula, disattivandole chimicamente prima che possano interagire con il ribosoma.
I ricercatori hanno pensato che ci doveva essere un modo per apportare modifiche alla streptogramine tale da renderle invisibili alle Vats.
Per testare questa ipotesi, optarono per una sintesi ex novo di varianti di streptogramine invece di usare il metodo classico di apportare modifiche alla struttura preesistente. Per rendere più semplice il processo di sintesi e modifica, la molecola venne "divisa" in sette moduli molecolari (modificabili separatamente) assemblabili facilmente in modo da ottenere varianti dell'antibiotico.
Ottenuti i "mattoncini" costitutivi, il passaggio chiave è stato quello di sviluppare un modo per visualizzare l'effetto di ciascuna modifica sulla capacità di legare il ribosoma e di sfuggire alle Vats, evitando nel contempo test su varianti "inutili". A tale scopo si avvalsero delle informazioni ottenute da tecniche come la microscopia crioelettronica e la cristallografia a raggi X, con cui creare immagini 3D a risoluzione quasi atomica del farmaco, del suo bersaglio e della sua nemesi (la proteina Vat).
E' stato grazie a questa modellistica molecolare che i ricercatori hanno potuto prevedere quali parti della molecola di streptogramina erano essenziali perché continuasse ad esercitare la funzione di antibiotico e quali erano quelle su cui agire per impedire alle Vats di interagire con il farmaco. Dei sette mattoncini costitutivi usati, due si sono rivelati i migliori substrati su cui effettuare le modifiche.
Le streptogramine varianti così selezionate sono state infine testate contro batteri patogeni prima (test di attività) e, quelle che avevano superato il test, contro il ceppo di S. aureus resistente alla streptogramina inoculati in topi. Alcune delle varianti così testate si sono rivelate 10 volte più efficaci delle molecole di partenza nel neutralizzare i batteri (un processo che può continuare fino ad ottenere l'attività desiderata).
L'approccio non è ovviamente limitato alle sole streptogramine ma, con le opportune correzioni dei mattoncini di partenza, utilizzabile su qualunque altro antibiotico "perso per strada".
Questa intuizione sarà una guida per creare antibiotici a cui i batteri non possono resistere ... o almeno non a breve termine
Altri articoli tematici li trovate seguendo il tag

-->antibiotici e -->superbatteri
Fonte
- Synthetic group A streptogramin antibiotics that overcome Vat resistance
***.
In questa lotta potremo avere alleati inattesi, i virus batterici. Organismi perfetti come arme antibatteriche in quanto mirate a ceppi particolari e in grado di coevolvere autonomamente per superare le contromisure di resistenza adottate dai loro bersagli, in una lotta che va avanti dall'inizio dei tempi sulla Terra.
Un esempio delle potenzialità insite nei batteriofagi, il loro utilizzo sul campo per il trattamento di una infezione intrattabile che aveva colpito il marito di una ricercatrice durante un viaggio in Egitto. Il resoconto delle procedure messe in atto è mirabilmente descritto nel libro "The Perfect Predator" (di Strathdee & Patterson) scritto in modo comprensibile a tutti.



Nessun commento:
Posta un commento