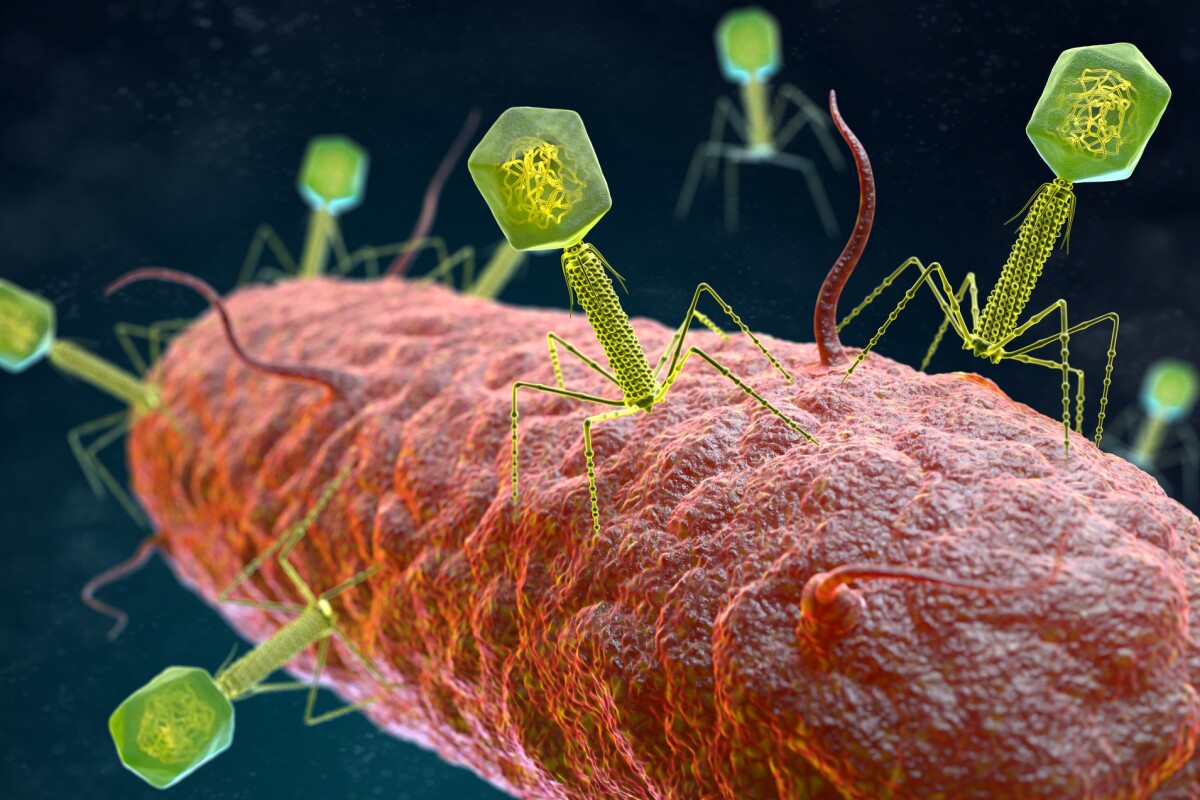
La creazione di batteri resistenti a qualunque virus
Tali virus sono per noi del tutto inoffensivi per una ragione intrinseca al loro funzionamento che giustifica la
definizione di quasi-organismi: sono paragonabili a sequenze di codice operativo che necessitano di un macchinario preesistente, la cellula. Da soli non sono in grado di fare nulla se non rimanere inerti come granelli di sabbia.
I virus non solo necessitano di una cellula ma di una tipologia ben particolare di cellula all'interno di un organismo, come ad esempio quelle dell'epitelio respiratorio. La ragione è nella loro dipendenza dalla "porta d'ingresso", il recettore, a cui il virus deve agganciarsi per riuscire ad entrare (attivando sistemi automatici cellulari o nel caso di virus dotati di
pericapside sfruttando la sola prossimità della membrana). Questo potrebbe anche non bastare, perché alcuni virus (ad es. i retrovirus) necessitano di una cellula in divisione per poterne sfruttare il macchinario replicativo (ad es. i neuroni sarebbero un bersaglio "inutile" anche una volta penetrati nella cellula, essendo in
fase G0).
L'insieme di questi limiti spiega perché pur vivendo noi in un ambiente pieno di virus, siamo per il 99,99% di essi assolutamente invisibili. Per rendere il concetto è come se il virus avesse il manuale per dirottare l'operatività cellulare per i suoi fini ma per eseguire le istruzioni non solo deve trovare un "veicolo" compatibile ma anche parlare la "stessa lingua". In caso contrario il genoma del virus penetrato nella cellula rimarrebbe inerte fino ad essere distrutto dagli enzimi cellulari come "spazzatura". A volte c'è un certo grado di "tolleranza" tra le "lingue" e questo spiega le infezioni derivanti da zoonosi che danno luogo ad infezioni "difettose" da cui, a volte, possono scaturire virus mutati più adatti al nuovo ospite.
Chiaramente nel caso di virus procariotici (il cui bersaglio sono
eubatteri o Archea) non abbiamo la complessità e variabilità dei bersagli tipici di un organismo pluricellulare. Un dato virus infetta cellule di una o più specie batteriche purché queste siano permissive a livello di recettore e di "macchinario cellulare" e non abbiano evoluto meccanismi di resistenza per quel dato virus.
Sarebbe possibile immaginare di rendere una data cellula (per semplicità limitiamoci ai soli batteri) "invisibile" ai virus ambientali?
La strategia più semplice sarebbe eliminare il/i recettore/i riconosciuti dal virus.
Fattibile mediante ingegneria genetica ma non sempre possibile senza alterare (o perfino uccidere) la cellula; il recettore usato dal virus non è usato, ovviamente, dalla cellula per fare un favore al loro mortale nemico ma perché adempie a funzioni specifiche come l'interazione con molecole esterne; è il virus ad essersi evoluto producendo sulla propria superficie la "chiave di ingresso" riconosciuta dal recettore.
C'è un altro modo ipotizzato da anni ma impossibile da mettere in pratica anche solo 5 anni fa, data la mole di lavoro necessaria con gli strumenti di ingegneria genetica allora disponibili.
Si tratta di cambiare il sistema di decodifica dell'informazione genetica della cellula pur mantenendo la stessa strutturalmente e funzionalmente identica alla versione non modificata.
Per capire il senso dell'approccio bisogna spendere alcune righe sul codice genetico.
L'informazione genetica è racchiusa, nella forma più semplice e diretta quale quella batterica, in una sequenza di nucleotidi nel DNA che viene poi copiato sotto forma di RNA, la cui informazione viene poi tradotta in una sequenza di aminoacidi da un complesso ribonucleoproteico fatto di ribosomi e tRNA. L'informazione nella sequenza dell'acido nucleico è veicolata sotto forma di "bit" fatti da 3 nucleotidi (tripletta) che definisce un codone a cui corrisponde, una volta tradotto, un particolare aminoacido.
Data una ipotetica sequenza di 300 nucleotidi sul mRNA a partire da un segnale di "inizio lettura", questa verrà scansionata da un "lettore" che si muove a blocchi di 3 nucleotidi adiacenti alla volta, e letta come una sequenza di 100 codoni. In corrispondenza di ciascun codone verrà aggiunto alla proteina in fase di sintesi l'aminoacido corrispondente a quel codone. Il risultato finale è una proteina di 100 aminoacidi.
Poiché i nucleotidi sono 4 (A, C, G, T o U nel mRNA) il numero di triplette possibili è 43=64.
Poiché il numero di aminoacidi codificati è 20 vuol dire che il codice è ridondante (detto anche degenerato). Anche dopo avere rimosso le 3 triplette di STOP, che segnalano "fine lettura", arriviamo a 61 codoni utili; la ridondanza è data dal fatto che alcuni aminoacidi sono codificati da un solo codone mentre altri da più codoni (ad esempio la valina da 4 codoni, la serina da 6, etc).
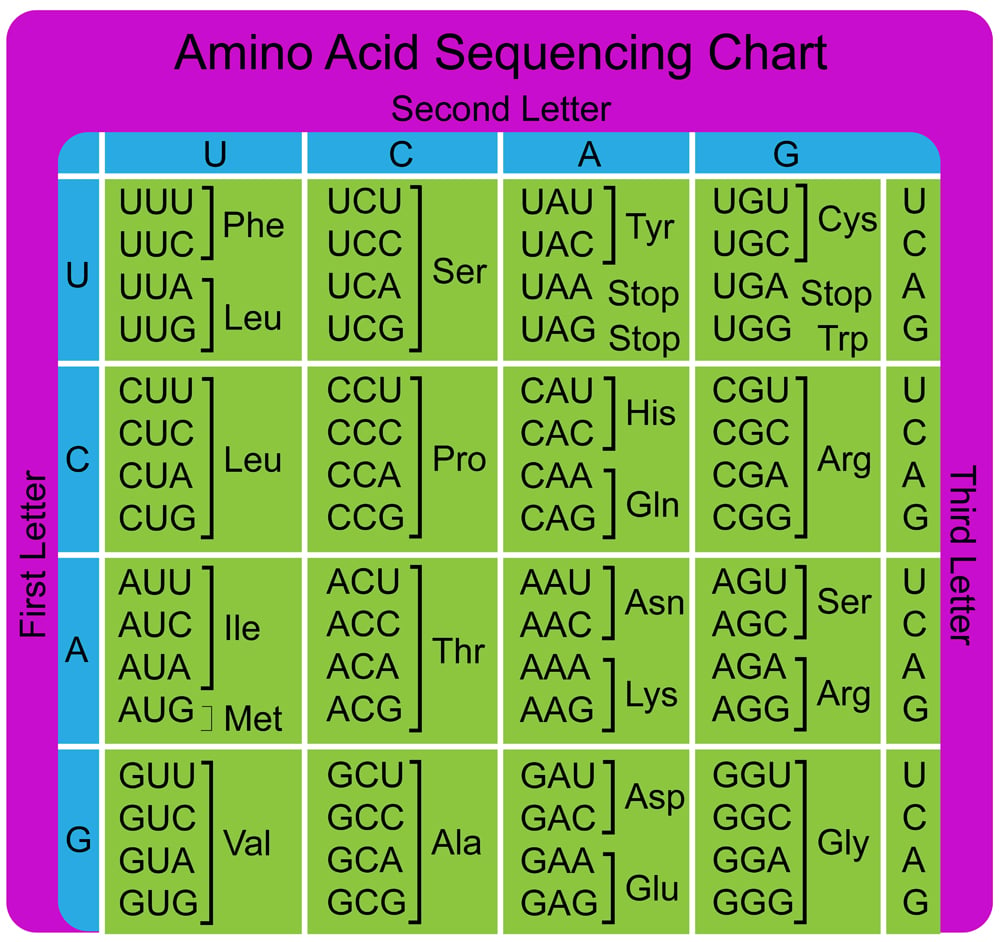 |
| La tabella di conversione tra la tripletta sul RNA (codone) e l'aminoacido "montato" dal ribosoma durante la lettura del mRNA. Si parla di codice degenerato perché alcuni aminoacidi sono codificati da più triplette. Per una buona descrizione, semplificata quel tanto che basta, del processo traduzionale vi rimando al sito scienceabc.com |
Nella realtà dei fatti la maggior parte degli organismi non si cura di più di 50 codoni come dimostrato dal possedere al massimo 50 tRNA specifici (i decodificatori del codone): una decina di codoni non sono mai quindi mai presenti nell'informazione genica senza che questo abbia alcun effetto sull'effettivo utilizzo di tutti e 20 gli aminoacidi naturali. Tale predilezione per alcuni codoni si è evoluta in modo diverso nei vari organismi tanto che quando si vuole far esprimere un gene esogeno in una cellula di un organismo terzo è buona norma procedere alla "ottimizzazione" dei codoni privilegiando quelli che l'organismo usa di più (ad esempio selezionando 1 delle 4 triplette possibili quando c'è l'informazione per la valina).
Poiché i virus utilizzano il sistema di decodifica della cellula, avranno verosimilmente ottimizzato i codoni in uso in accordo con la cellula bersaglio.
Quest'ultimo aspetto ci fornisce la chiave per comprendere la strategia usata dai ricercatori nel creare un batterio resistente ad ogni virus, anche a quelli capaci di penetrare nella cellula.
Il termine "sintetico" indica che il DNA (lungo 4 milioni di nucleotidi) è stato interamente sintetizzato in vitro usando come riferimento la versione naturale del genoma e poi inserito, pezzo a pezzo al posto del DNA originale in una preesistente cellula batterica.
Il batterio sintetico risultante era strutturalmente e funzionalmente uguale al "genitore", come ovvio dato che nessuna alterazione "post-codifica" era stata introdotta.
Un vero tour de force impensabile come detto anche solo un quinquennio fa ma oggi praticabile, per quanto laborioso e tedioso (in tutta franchezza non invidio in nulla i ricercatori).
In poche parole i ricercatori hanno modificato 3 dei 20 codoni naturali (per un totale di 18 mila codoni), cambiandone "destinazione d'uso": invece di codificare per i classici aminoacidi sono stati "associati" ad aminoacidi non esistenti in natura.
Il concetto può essere semplificato prendendo in esame l'aminoacido serina: se il codone TCG è sufficiente per l'inserimento della serina nella proteina nascente, perché non editare gli altri 3 codoni serinici (TCA, AGC e AGT) trasformandoli in TCG? La proteina prodotta sarebbe esattamente identica con il vantaggio di avere "liberato" tre codoni. Questi potrebbero sia essere lasciati vacanti (vedi sotto) o anche usati per creare proteine con aminoacidi disegnati a tavolino.
Tra il dire e il fare c'è di mezzo "riprogrammare" l'intero apparato di traduzione riassumibile in questi passaggi:
- modificare TUTTE le triplette "in eccesso" convertendole in (vedi esempio precedente) TCG;
- modificare i geni codificanti per i tRNA che riconoscono le tre triplette ex-serina dissociandole da questo aminoacido e, volendo, associandole ad un aminoacido sintetico.
- (opzionale) modificare le vie biosintetiche della cellula affinché diventi capace di sintetizzare il "nuovo" aminoacido. Passaggio in realtà non necessario se l'idea è quella di fornire l'aminoacido sintetico nel brodo di coltura ottenendo così il vantaggio di un organismo incapace di produrre le "nuove" proteine, e magari non sopravvivere, al di fuori dell'ambiente di laboratorio o di un ambiente in cui tali aminoacidi siano forniti insieme al cibo.
Al termine di questo editing certosino avremo un genoma sintetico che, prendendo in esame la sola versione di modifica minimale (eliminazione dell'associazione tra i 3 tRNA (TCA, AGC e AGT) con la serina renderà questo organismo "letale" per qualunque virus voglia usarlo per replicarsi una volta penetrato nella cellula.
La ragione è semplice: il genoma virale prevede ancora che in corrispondenzza dei codoni TCA, AGC e AGT venga inserita nella proteina virale una serina; tuttavia il macchinario cellulare interpreta questi codoni in modo del tutto diverso inserendo il nuovo aminoacido oppure, se i tRNA dedicati sono stati rimossi in fase di editing), lasciando uno spazio non riempibile. Risultato, il blocco immediato della sintesi proteica che attiene alla decodifica del solo mRNA virale.
In un certo senso possiamo pensare l'evento come al cambio di linguaggio macchina a cui nessun virus può adeguarsi per quanto alto sia il suo tasso di mutazione: dovrebbe mutare tutti e tre i codoni "inutili" in tutto il genoma virale e nello stesso momento. Impossibile.
La teoria è stata messa alla prova aggiungendo al terreno di coltura di questi nuovi batteri, noti come Syn61.Δ3(ev5), un mix di virus tutti capaci di infettare in modo produttivo (leggasi produrre nuovi virus) il batterio originario. Nessuna infezione ha avuto esito positivo, sebbene il virus non abbia avuto problemi nel penetrare la cellula, e con ciò la dimostrazione di un batterio resistente a qualunque virus naturale.
Oltre alla valenza scientifica intrinseca di questo approccio, si può pensare a futuri utilizzi di batteri sintetici (organismi più complessi sono fuori dalla portata tecnologica se non con modifiche molto mirate) per compiti tra i più disparati: mini-fabbriche facilmente riprogrammabili utilizzabili sia nella produzione di farmaci che per la bonifica di aree contaminate attraverso la sintesi di proteine di nuovo tipo grazie all'utilizzo di "mattoni" disegnati ad hoc.