L'articolo odierno è un poco specialistico ma cercherò di semplificarlo al massimo in modo che arrivi a tutti la potenza intrinseca al metodo descritto.
Parlo di biologia molecolare e di una tecnica pubblicata poche settimane fa, nota come SLIDE-SEQ, con la quale è possibile ricostruire l'identità e la posizione di ogni singola cellula in un tessuto.
Nota. La SLIDE-SEQ è una variante della tecnica nota come RNA-SEQ.
L'analisi al microscopio ottico, grazie all'utilizzo di opportune colorazioni e alla morfologia cellulare, permette un certo grado di riconoscibilità e qui entra in gioco l'esperienza dell'istopatologo capace di riconoscere la tipologia cellulare ed eventuali anomalie strutturali del tessuto.
Quando da un insieme tutto sommato "semplice" come quello del polmone si passa al cervello la complessità cresce in modo esponenziale sia per l'aumento sia della tipologia di cellule che della organizzazione 3D e della rete di connessioni, con variazioni importanti anche tra aree distanti meno di 1 cm. In questo ambito non è importante distinguere solo un neurone da un astrocita (tralasciando le pur importanti cellule che delimitano i vasi sanguigni) ma è fondamentale capire la tipologia del neurone, ad esempio un neurone dopaminergico da uno che libera GABA. Anche qui la morfologia aiuta (vedere i bellissimi neuroni cerebellari del Purkinje, tipicamente GABAergici, presenti nel cervelletto), altre volte la identificazione visiva è quantomeno improba.
 |
| Cellule del Purkinje (credit: BrainsRusDC via wikipedia) |
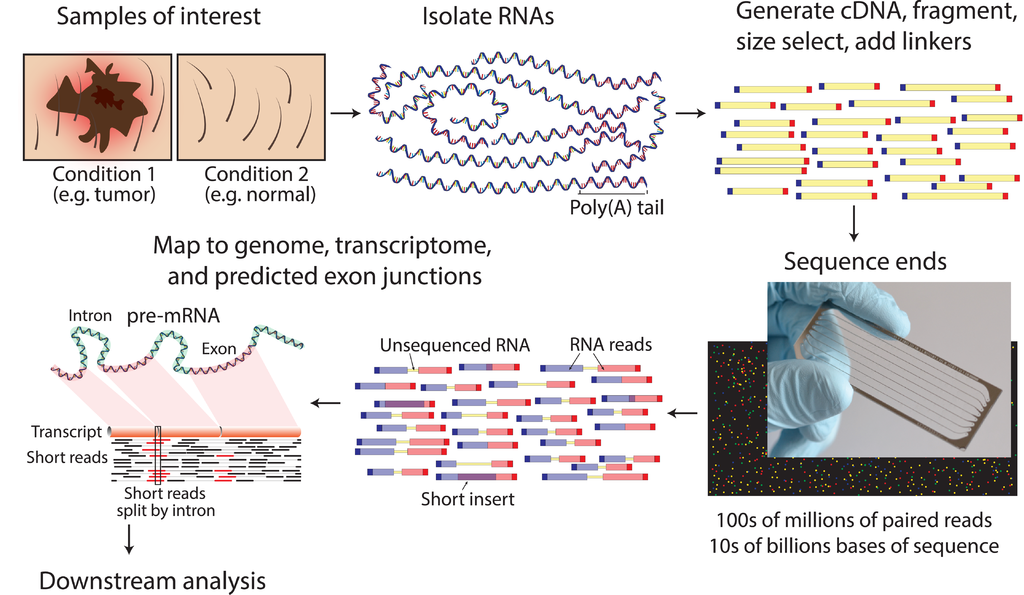
Negli ultimi anni le tecniche si sono affinate a tal punto da permettere di identificare una cellula in base al tipo di espressione genica, riconducibile al tipo di RNA messaggeri prodotti. La identificazione quali-quantitativa del tipo di mRNA presenti si basa su tecniche come retrotrascrizione (RNA come stampo per generare DNA) e sequenziamento, il cui risultato è una "impronta digitale" di una cellula in un dato stadio di maturazione.
Parliamo di "impronte" risultato della valutazione di migliaia di messaggeri diversi, che a volte possono differire in modo sostanziale per la presenza/assenza di una o poche specie. Un lavoro che oggi è gestito "facilmente" grazie alla capacità elaborativa dei computer che definiscono un pattern di espressione analizzando in parallelo migliaia di campioni diversi provenienti (idealmente) da cellule singole
E' possibile associare gli RNA prodotti da una particolare cellula (e solo da quella) etichettando tutte queste molecole con un barcode nel momento in cui la cellula viene rotta in prossima di un "sensore" di RNA.
Ottenuta l'impronta "funzionale", questa viene comparata con quelle presenti in database relative a cellule già "catalogate", ottenendo così l'identificazione di quale cellula si trova in una data posizione.
Vediamo a grandi linee quello che hanno fatto i ricercatori del team di Evan Macosko i cui laboratori si trovano al Broad Institute (Cambridge, USA).
Nota. Gli stessi autori che qualche hanno fa avevano sviluppato un'altra tecnica molto interessante nota come DROP-SEQ.
Su un vetrino opportunamente trattato vengono posizionate migliaia di "beads" (palline sintetiche) ricoperte di piccoli e molteplici frammenti di DNA necessari per "catturare", appaiandosi, con l'RNA con cui vengono a contatto. Ciascuna pallina è diversa in quanto i frammenti di DNA sulla sua superificie sono univocamente "taggati" con un barcode, diverso in ciascuna pallina.
Le palline, fissate al vetro, hanno un diametro di circa 10 uM, di poco inferiore alla dimensione media di una cellula. Dimensione non casuale: ciascuna pallina interagirà con molta probabilità con una singola cellula.
Prima di iniziare l'esperimento la posizione di ciascuna pallina sul vetrino viene mappata così da avere una indicazione univoca della posizione di un dato segnale alla fine dell'esperimento.
L'esperimento vero e proprio inizia prelevando una piccola porzione del tessuto in esame, che ovviamente è tridimensionale, per affettarla (come farebbe un salumiere ma con "fettine" ampie pochi mm e spesse qualche micron). Ciascuna fettina viene prelevata e posizionata su singoli vetrini fino ad esaurimento del campione. Una volta a contatto con il vetro la cellula viene lisata in modo che il suo contenuto fuoriesca ed entri in contatto con le palline immediatamente adiacenti.
Dopo una serie di lavaggi si procede ad amplificare il numero di copie di RNA associati alle palline: per prima cosa le si "retro-trascrive" a formare DNA (complementare al RNA di partenza) e infine si amplificare il numero di frammenti in modo che possano essere facilmente quantificati.
Ricordo che ciascuno di questi frammenti si porta dietro un barcode unico, riconducibile alla pallina di origine, mappata all'inizio.
 |
| Visualizzazione dei passaggi chiave della tecnica. Posizionamento delle beads sul vetrino, mappaggio, aggiunta sezione del tessuto, processamento del campione. (Credit: Makosko Lab / sciencemag.org (crop from original pict --> here) |
Gli autori hanno testato la procedura sull'ippocampo una particolare area del cervello fondamentale per la formazione della memoria. Il risultato finale è nella GIF sottostante.
Se vi chiedete dove sia localizzato l'ippocampo (anzi "gli", essendo 2, uno per emisfero), vi do un aiuto visivo
Fonti
- Evan Macosko lab
- Slide-seq: A scalable technology for measuring genome-wide expression at high spatial resolution
 |
| Le informazioni raccolte su ciascuna fettina di tessuto permette di ottenere una sorta di "GPS" funzionale dell'ippocampo (credit. Macosko labs) |
Se vi chiedete dove sia localizzato l'ippocampo (anzi "gli", essendo 2, uno per emisfero), vi do un aiuto visivo
 |
| credit: Life Science Databases(LSDB) |
Se avete trovato interessante la tematica vi rimando a due articoli di questo blog in cui ho sommariamente descritto tecniche visivamente spettacolari come --> CLARITY e --> BRAINBOW
Fonti
- Evan Macosko lab
- Slide-seq: A scalable technology for measuring genome-wide expression at high spatial resolution
Samuel G. Rodriques et al, (2019) Science, Vol. 363, Issue 6434, pp. 1463-1467
- A GPS-like System for Single-Cell Analysis
Francis Collins blog (director of NIH)
- A GPS-like System for Single-Cell Analysis
Francis Collins blog (director of NIH)

Nessun commento:
Posta un commento