I cultori di serie TV come CSI reputano prassi consolidata le tecniche di indagine fornite dalla genetica forense, grazie alle quali il tecnico di laboratorio di turno fornisce all'investigatore la chiave per identificare il reo.
In verità molto di quello che viene mostrato è pura bischeraggine (giusto per usare un eufemismo), specie quando attiene ai tempi necessari per fare una analisi, la strumentazione usata e soprattutto il multitasking del tecnico capace di usare ugualmente bene, e nello stesso laboratorio, uno spettrometro di massa e un sequenziatore next-gen. Ma si tratta di puro intrattenimento ed è bene sottolinearlo per evitare le false aspettative in coloro che poi "pretenderanno" di vedere in azione queste procedure nelle indagini reali, stupendosi poi della lentezza e magari della mancanza di risultati.
In verità molto di quello che viene mostrato è pura bischeraggine (giusto per usare un eufemismo), specie quando attiene ai tempi necessari per fare una analisi, la strumentazione usata e soprattutto il multitasking del tecnico capace di usare ugualmente bene, e nello stesso laboratorio, uno spettrometro di massa e un sequenziatore next-gen. Ma si tratta di puro intrattenimento ed è bene sottolinearlo per evitare le false aspettative in coloro che poi "pretenderanno" di vedere in azione queste procedure nelle indagini reali, stupendosi poi della lentezza e magari della mancanza di risultati.
Con questo non si vuole sottostimare la potenzialità della tecnologia forense ma solo contestualizzarla alla realtà delle procedure (e dei tempi) nel mondo reale.
In Italia i reparti della scientifica (ad esempio i RIS) hanno ben poco da invidiare ai colleghi d'oltreoceano come ben evidenzia il lavoro immane di ricostruzione del profilo genetico nel caso dell'omicidio di Yara Gambirasio.
Rimanendo nell'ambito della genetica forense possiamo delineare due metodiche di indagine: la ricerca della concordanza genetica tra il reperto trovato sul luogo del crimine e il campione biologico di un indiziato; la ricostruzione del profilo dell'indiziato "ignoto" dal DNA trovato, in assenza di una lista di sospettati o qualora questi abbiano un profilo genetico non compatibile con la traccia.
Il primo caso non necessita di ulteriori commenti essendo un tormentone di qualunque serie poliziesca. Se la concordanza tra i marcatori genetici supera una certa soglia statistica, per cui è "improbabile essere dovuta al caso" (la statistica ragiona in questi termini), allora il riscontro avrà carattere probatorio.
Il secondo caso, di cui scriverò oggi, è più variegato e quindi di interesse, perché incorpora sia tecniche oggi routinarie che potenzialità un tempo fantascientifiche ma oggi prossime all'utilizzo nel mondo "reale".
Il secondo caso, di cui scriverò oggi, è più variegato e quindi di interesse, perché incorpora sia tecniche oggi routinarie che potenzialità un tempo fantascientifiche ma oggi prossime all'utilizzo nel mondo "reale".
Nessuno si stupirebbe troppo oggi nell'apprendere che è possibile risalire, da un campione biologico anche minimale, non solo al sesso ma anche all'etnia e perfino all'età di chi ha lasciato la traccia. Tutto dipende sia dall'abbondanza che dallo stato di conservazione del campione, che deve consentire la rilevazione di "impronte digitali" genetiche (oltre a tracce chimiche di altra natura) che altro non sono che sequenze nucleotidiche (il DNA) associate ad un particolare carattere.
Ad esempio le rilevazione di sequenze di DNA appartenenti al cromosoma Y indicano che il "proprietario" della traccia è un maschio così come altri marcatori genetici ci potranno dire se è biondo o se è africano, se è un nano o se è alto. Esempi non casuali perché è molto più semplice identificare due estremi invece della media della popolazione dove le differenze, pur presenti, necessitano di molti più marcatori per essere identificati. Non spaventatevi se vi appare nebuloso, lo capirete meglio quando spiegherò "quanta informazione" ci vuole per determinare caratteristiche esterne banali come il colore dei capelli, etc.
In un futuro dietro l'angolo, o che forse è già qui, il DNA da solo potrebbe fornire un identikit sufficientemente dettagliato da essere usato come foto segnaletica.
Una possibilità che è il sogno di ogni dipartimento di polizia. Raccogliere tracce di DNA da una scena del crimine, inserirle in un dispositivo portatile e da questo generare nel tempo di un caffè l'istantanea di chi ha lasciato queste tracce.
Una possibilità che è il sogno di ogni dipartimento di polizia. Raccogliere tracce di DNA da una scena del crimine, inserirle in un dispositivo portatile e da questo generare nel tempo di un caffè l'istantanea di chi ha lasciato queste tracce.
Alcune persone, non disinteressate, vorrebbero far credere che quel giorno è già arrivato. Persone, giusto per fare dei nomi, come gli autori di un articolo pubblicato sulla rivista PNAS che sostenevano di aver sviluppato un algoritmo capace di predire correttamente l'aspetto di una persona, partendo dal genotipo (non sempre ma il 74% delle volte … ). Articolo che ha sollevato critiche circa la faciloneria di alcune sue affermazioni che sembravano veicolate più da futuri interessi commerciali che dalla fattibilità e affidabilità del risultato. Il punto infatti non è se sia possibile ricavare informazioni sul "proprietario" del DNA (questo è possibile senza dubbio) ma nel definire la tecnica come matura a sufficienza per essere usata da sola come prova,
Nota. Gli autori lavorano per una biotech, la Human Longevity, fondata da Craig Venter (l'artefice del sequenziamento del DNA umano fatto un consorzio privato che si contrappose al Progetto Genoma pubblico)
Molto più affidabile e degno di citazione il lavoro di un team della Columbia University che è riuscito, partendo dal DNA ricavato dalle tracce biologiche e con un analizzatore portatile, a validare algoritmi predittivi delle caratteristiche dei soggetti. Vero che gli autori non fanno affermazioni circa l'utilizzo del metodo in ambito forense, ma si tratta di una applicazione che è di fatto "dietro l'angolo" una volta superate le forche caudine della verifica su larga scala.
Al momento è più appropriato parlare di fotofitting molecolare, cioè la predizione del fenotipo (tra cui la morfologia) partendo dal DNA, un approccio che fa già parte degli strumenti forensi. In USA ci sono aziende specializzate in queste ricostruzioni, come la Parabon, che per qualche migliaio di dollari forniscono agli inquirenti un identikit del "proprietario" della traccia biologica, grazie all'utilizzo di una ampia collezione di varianti genetiche (polimorfismi noti come SNP) associati a tratti morfologici specifici. Un prezzo accettabile da pagare per risolvere cold cases vecchi di decenni.
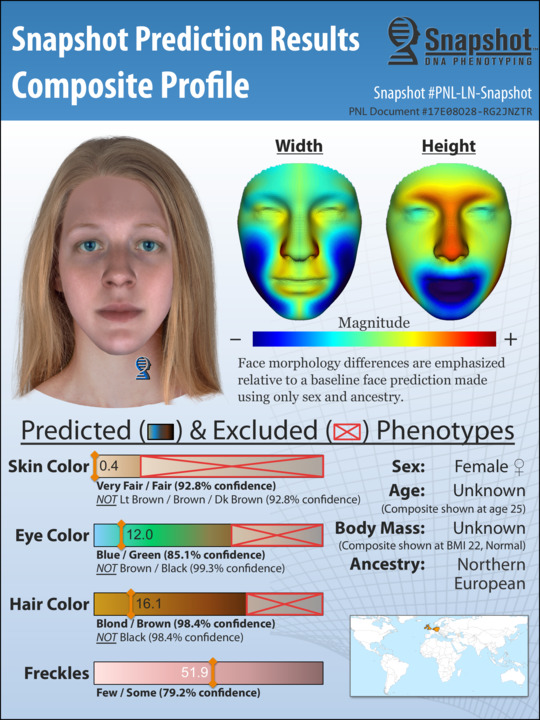
Un esempio della capacità predittiva dell'algoritmo, tratto dal sito della Parabon.
Un esempio della capacità predittiva dell'algoritmo, tratto dal sito della Parabon.
Il procedimento in sé non ha nulla di "fantascientifico", facile da capire per chiunque abbia anche solo una minima pratica di genetica molecolare. La chiave di tutto è uno (o più) chip su cui sono depositate le informazioni genetiche di cui si vuole valutare la presenza nel campione in esame.
Questi chip nulla hanno a che fare con i chip silicei usati nei dispositivi elettronici. I chip usati in biologia molecolare sono dei piccoli supporti suddivisi in minuscole celle in cui sono localizzati (ad esempio) stringhe di una determinata sequenza di DNA, che definisce un dato polimorfismo. I chip vengono messi a contatto con il DNA estratto (processato e spesso amplificato) dal campione in modo che le sequenze identiche (o meglio complementari) possano appaiarsi. Passaggio finale sarà la scansione del chip per identificare in quali celle è avvenuto l'appaiamento e quindi quali sono i marcatori che andranno considerati nella fase di profilazione.
 |
| Esempio generico di un DNA chip usato per monitorare l'espressione dei geni in un determinato tessuto. |
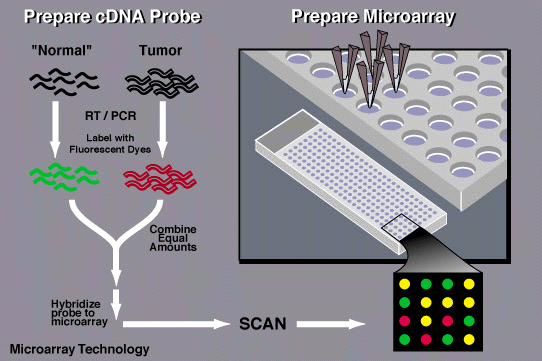 |
| Una delle applicazioni classiche in oncologia molecolare è la comparazione dell'attività genica tra cellule normali e tumorali in un dato tessuto. In modo concettualmente analogo viene fatta la comparazione tra il DNA di diversi campioni per verificarne la somiglianza (image credit: NIH) |
La scarsa qualità dei campioni viene "pesata" da un algoritmo interno che ricalcola i risultati rimuovendo le informazioni troppo "deboli" e che quindi si comporterebbero come outlier.
Il risultato finale prevede l'intervento umano sotto forma degli "artisti forensi" i quali traducono l'insieme delle previsioni morfologiche fatte dal DNA in uno snapshot (istantanea) dell'indiziato.
Chiaramente lo snapshot non è paragonabile ad un identik fatto da un testimone oculare (fatto salvo che da tempo è nota la scarsa affidabilità dei ricordi). Non potrà prevedere caratteristiche non genetiche come il naso schiacciato di un ex-pugile oppure una cicatrice o alterazioni estetiche volute (dai tatuaggi al trapianto di capelli in un soggetto "predetto" come calvo) ma si è già dimostrata utile in più di una indagine.
Tra queste quella di un sospetto che ha confessato l'omicidio dopo aver visto lo snapshot (il suo) ricavato dal DNA lasciato sulla scena del delitto a Dallas.
Chiaramente lo snapshot non è paragonabile ad un identik fatto da un testimone oculare (fatto salvo che da tempo è nota la scarsa affidabilità dei ricordi). Non potrà prevedere caratteristiche non genetiche come il naso schiacciato di un ex-pugile oppure una cicatrice o alterazioni estetiche volute (dai tatuaggi al trapianto di capelli in un soggetto "predetto" come calvo) ma si è già dimostrata utile in più di una indagine.
Tra queste quella di un sospetto che ha confessato l'omicidio dopo aver visto lo snapshot (il suo) ricavato dal DNA lasciato sulla scena del delitto a Dallas.
Metodi e dettagli tecnici sono secretati al di fuori dell'ambito processuale. Semmai si è in più riprese contestata la liceità del prelievo del DNA, in assenza di esplicita autorizzazione dell'indiziato anche nel corso di indagini ufficiali. In quasi tutti gli stati USA la legge definisce come ammissibile l'utilizzo di tracce biologiche lasciate "spontaneamente" da un individuo come può essere il caso della saliva lasciata sul bicchiere usato al bar oppure capelli persi. Parla molto chiaro in tal senso una sentenza della Corte Suprema in seguito al caso King vs. Maryland:
"In the case of surreptitious collection of DNA, the argument rests on the notion that the DNA has been voluntarily abandoned, and does not have the same legal status as property". Quindi non esiste il reato di "furto di DNA" se il DNA è stato abbandonato.
Il problema maggiore nella ricostruzione fenotipica del "possessore" della traccia biologica (potrebbe infatti appartenere sia alla vittima di cui non si trova il corpo o al perpetratore del crimine) è che esistono tratti "facili" (colore pelle, occhi, capelli) e altri ben più complicati come la fisionomia del volto. Non a caso gli stessi artisti forensi spiegano che i volti da loro prodotti sono stereotipati, generiche rappresentazioni di possessori di alcune caratteristiche e non veri identitik ottenuti da un testimone. Utili perché evidenziano caratteristiche somatiche che possono escludere qualcuno o all'inverso focalizzare l'attenzione su un soggetto altrimenti non considerato.
Parlare di tecnologia di riconoscimento facciale basata sul DNA in senso stretto è quindi prematuro. Soprattutto considerando che sesso, ascendenza, età e anche la dimensione del corpo contribuiscono tutti a definire la morfologia del viso.
Sesso e ascendenza contribuiscono per il 25% mentre un altro 20% che determina la forma del volto è il risultato di almeno 16 varianti.
Meglio definirla allora come un ausilio investigativo, utile quanto basta perché osservatori umani (con familiarità per la popolazione in esame) riconoscano le piccole differenze che definiscono un volto tra tanti osservati.
Credo sia utile, come nota finale, fare un breve accenno alla genetica che determina i tratti più evidenti della fisionomia (colore capelli, occhi, pelle e altezza), per comprendere quanto la determinazione di un carattere possa essere praticamente certa o probabilistica a seconda di quale sia il tratto somatico valutato.
Colore occhi
I geni coinvolti nel colore dell'iride sono 6, quindi la capacità previsionale è abbastanza buona. Da qui in poi userò una scala metrica dove 0,5 indica predizione casuale mentre 1 è predizione sicura. Senza addentrarci nella tipologia dei geni coinvolti possiamo dire che la predizione del colore blu, marrone e intermedio è dello 0,94 , 0,95 e 0,74 rispettivamente. Vale la pena precisare che varianti cromatiche "simili" come blu, verde e azzurro sono spiegabili dell'effetto Tyndall (simile a quanto avviene per il colore del cielo a causa dello scattering di Rayleigh) nello stroma dell'iride. Il colore chiaro dell'iride non dipende infatti dalla presenza di pigmenti verdi o blu ma sono entrambe conseguenza della bassa concentrazione di melanina nell'iride. Se ad alte concentrazioni questa determina il colore marrone o nero, a basse concentrazioni da luogo ai colori di cui sopra (con tonalità che possono variare a seconda dell'incidenza della luce). La tendenza al verde è dovuta alla presenza di un pigmento giallo (lipocromo) che sommato all'effetto blu dovuto alla dispersione della luce nello stroma genera il verde.
Capelli
Il colore dei capelli (chiaramente quello naturale) è un poco più complicato perché è condizionato dall'età. Credo che tutti abbiate notato come le bionde naturali diventino castane già dopo l'adolescenza per poi virare verso il bianco. Cosa che non avviene con chi ha di partenza i capelli castani. Pertanto, le previsioni sui capelli castani sono meno accurate (0,75) rispetto ai capelli biondi (0,81), neri (0,92) e rossi (0,93). Avrete notato che il colore rosso è il più "facile" da prevedere; la ragione è nel determinante genetico, allele recessivo del gene MC1R che si trova sul cromosoma 16; se trovate due copie "concordi" dell'allele la determinazione di capello rosso è pressoché certa
La predizione genetica del colore dei capelli oggi in uso su basa sull'analisi di 24 varianti in 11 geni.
Colore pelle
Qui il set di geni da valutare per avere un tasso di predizione accettabile, comincia ad aumentare: servono 36 varianti da 17 geni. La capacità predittiva è alta se la scala di pigmentazione usata è su 3 categorie (chiaro, scuro e nero scuro), un poco più bassa se la categoria "chiara" viene ulteriormente frazionata a dare un totale di 5 categorie (molto chiaro, chiaro, intermedio, scuro e nero scuro).
L'affidabilità del risultato è 0,97 (chiaro), 0,83 (scuro) e 0,96 (nero scuro) nel primo caso, e 0,74 (molto chiaro), 0,72 (chiaro), 0,73 (intermedio), 0,87 (scuro) e 0,97 (nero scuro).
Quando si passa all'analisi di altri tratti le cose si complicano perché sono il risultato dell'azione combinata di un numero molto alto di geni (o meglio di varianti cioè alleli) ciascuno dei quali contribuisce in minima parte, per cui la presenza di un dato allele ha predittività in sé nulla e deve essere considerato solo insieme ad altri. La complessità di indagare la genetica di questi tratti è che se ogni allele ha un effetto basso il numero di individui da caratterizzare geneticamente aumenta esponenzialmente.
Altezza
Sebbene gli studi sui gemelli dimostrino che la componente genetica influisce per l'80%, fare una previsione dell'altezza è quanto mai complesso a causa del numero di geni in azione: per avere una previsione di 0,75 è necessario valutare almeno 180 alleli. Più facile la predizione degli estremi cioè i molto alti e i bassi (il nanismo e il gigantismo sono casi a parte, pur se intrinsecamente eterogenei, perché associabili a condizioni patologiche).
Il tema è un poco più complesso perché nella determinazione dell'altezza abbiamo a che fare con la "ereditarietà mancante" (missing heritability) che consiste nel fatto che la somma dei singoli contributi apportati da ciascuna delle centinaia di varianti geniche è inferiore al contributo calcolato dagli studi familiari. In pratica manca ancora qualche informazione. A risolvere il problema è stato un recente studio del Queensland Brain Institute che ha identificato il tassello mancante nell'indice di massa corporea (BMI). Lo studio ha preso in esame il genoma (6 miliardi di nucleotidi) di 21620 persone partendo dal presupposto che le persone sono in qualche modo legate tra loro - anche se in modo remoto - quindi il DNA può essere usato per calcolare gradi di parentela.
Supponiamo, per esempio, che una coppia di cugini di terzo grado abbia una altezza tra loro più simile a quella che ha una coppia di cugini di secondo grado in un'altra famiglia: questo vuol dire che i primi hanno in comune più alleli "altezza" di un certo tipo. L'analisi al computer ha consentito così di identificare alleli di interesse (noti per il loro coinvolgimento nel determinare il BMI).
Altre caratteristiche del viso che possono essere previste sono la presenza di lentiggini, la struttura dei capelli (ricci o dritti), lo spessore della barba, la forma del sopracciglio (...).
La calvizie precoce (anche molto prima dei 50 anni) può essere predetta con precisione di 0,74 mediante 14 varianti da 12 geni. Aggiungendo 200 varianti l'accuratezza sale fino a 0,78.
Età
Un parametro utile in molti casi giudiziari è la valutazione dell'età reale dell'individuo (fa la differenza tra punibilità totale o parziale soprattutto nel caso di soggetti senza documenti affidabili). Un metodo oramai classico si basa sulla determinazione della lunghezza dei telomeri cioè delle parti terminali dei cromosomi, fatte da sequenze ripetute, cruciali per preservare l'integrità dell'informazione ad ogni replicazione. Semplificando il tutto possiamo dire che le cellule "giovani" mantengono attivo un sistema che preserva la lunghezza dei telomeri; con il tempo il sistema si disattiva e i telomeri tendono ad accorciarsi provocando a loro volta l'attivazione dei sistemi di emergenza cellulare che portano alla senescenza cellulare (blocco proliferativo). Il sistema di controllo viene bellamente ignorato dalle cellule tumorali che riattivano il sistema di riparazione dei telomeri evitando così il blocco proliferativo.
La valutazione della lunghezza dei telomeri (o l'attività enzimatica dell'enzima preposto al loro mantenimento) in laboratorio non è complessa ma ci sono altri modi, anche più affidabili, per determinare l'età biologica.
Uno di questi è la valutazione dello stato di metilazione in alcune aree del genoma note come CpG islands. Nel lavoro prima citato fatto alla Columbia, i ricercatori hanno valutato lo stato di metilazione in 353 di queste regioni; l'informazione dell'età biologica ha una precisione inferiore ai 3 anni (vedi anche l'orologio di Horvath). Molto interessante il fatto che il test sia utilizzabile su qualunque tessuto, anche prenatale, il che sta ad indicare che è utilizzabile sia per determinare l'età che lo stadio di sviluppo. Non vi basta? L'analisi permette di determinare anche il tessuto d'origine e se il genoma della persona in esame abbia subito stress ambientali come quello legato all'essere un fumatore oppure dall'essere infettato da HIV (i ricercatori affermano che può prevedere quanti pacchetti di sigaretta al giorno abbia fumato il soggetto) .
Lo stato di metilazione non è ancora in uso per scopi forensi perché complicato e si sta lavorando per sviluppare qualcosa di più semplice.
L'analisi comparativa del DNA è già oggi centrale in molti procedimenti penali (400 mila solo negli USA) ed è destinato a crescere mano a mano che aumenterà il numero di soggetti il cui DNA (per trascorsi giudiziari) entri a fare parte dei database forensi.
Chiaramente questo approccio ha un limite importante in quanto permette di scoprire correlazioni unicamente tra i pregiudicati o all'interno di piccole popolazioni dove gli abitanti decidono di farsi esaminare in massa per agevolare le indagini nel caso di crimini efferati. Il DNA photofitting può colmare il gap.
Fonti
- Identification of individuals by trait prediction using whole-genome sequencing data
C. Lippert et al, (2017) PNAS, 14 (38) pp. 10166-10171
- Rapid re-identification of human samples using portable DNA sequencing
S. Zaaijer et al, eLife
- Parabon nanolabs
- DNA makes an appearance
Nature Biotechnology
Articoli su temi correlati che potrebbero interessarvi
--> DNA come memoria digitale del futuro?
--> DNA origami e nanorobot
Fonti
- Identification of individuals by trait prediction using whole-genome sequencing data
C. Lippert et al, (2017) PNAS, 14 (38) pp. 10166-10171
- Rapid re-identification of human samples using portable DNA sequencing
S. Zaaijer et al, eLife
- Parabon nanolabs
- DNA makes an appearance
Nature Biotechnology


Nessun commento:
Posta un commento