La crescita esponenziale dei dati generati in ambito scientifico ha un rovescio della medaglia, cioè come immagazzinarli in modo sicuro e duraturo.
Il solo CERN ad esempio ha prodotto più di 100 petabyte di dati che devono essere archiviati e resi accessibili non solo per l'immediato ma soprattutto per le future generazioni di ricercatori. Tuttavia i supporti standard per l'archiviazione di dati, come dischi ottici, dischi rigidi e nastri magnetici, hanno una durata variabile ma sempre nell'ordine di anni, se conservati adeguatamente.
Facile capire quindi l'importanza della ricerca di migliori tecnologie di archiviazione dei dati. Se dobbiamo cercare il meglio perché non guardare al miglior sistema di codifica e di preservazione dell'informazione di quello sviluppato dalla Natura circa 3,8 miliardi di anni fa con la comparsa della vita sotto forma di cellule? E se parliamo di cellule e di informazione codificata ci riferiamo al DNA (--> Nota 1 a fondo pagina).
Il DNA, la molecola che codifica le informazioni biologiche, ha il pregio di avere una notevole stabilità (può conservarsi per millenni) e la capacità di immagazzinare informazioni con alto rapporto informazione/densità (può codificare l'equivalente di petabytes di informazioni per grammo).
Lo spunto per l'odierno articolo viene da un lavoro pubblicato su Nature Biotechnology da Lee Organick e colleghi in cui si descrive un metodo per archiviare 200 megabyte di informazioni digitali sotto forma di DNA e di come recuperare tali informazioni senza errori "di lettura".
L'idea non è nuova.
Fin dagli anni '60, quando dopo la definizione della struttura del DNA da parte di Watson, Crick, Franklin e Wilkins i ricercatori scoprirono il modo con cui l'informazione era codificata (sappiamo oggi che quello era solo il primo livello), gli ingegneri informatici cominciarono a sognare di sfruttare tale sistema per immagazzinare i dati (anche analogici come il testo di un libro) codificandolo sotto forma di DNA (--> Nota 2)
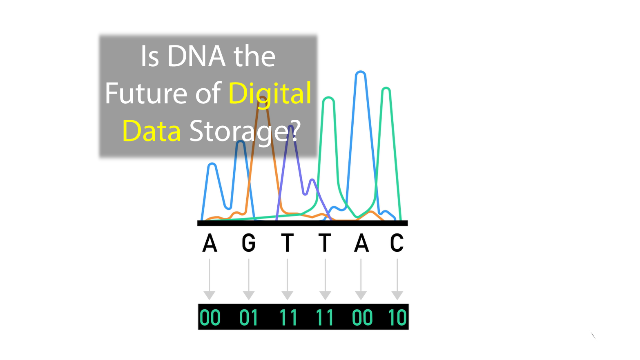
A prima vista, la memorizzazione delle informazioni nel DNA appare semplice. Le informazioni digitali, una serie di 0 e 1 nota come "bit", possono essere facilmente rappresentate come una combinazione di nucleotidi. Ad esempio la sequenza 00-01-10-11 potrebbe essere definita dalla sequenza A-C-G-T, facilmente sintetizzabile.
Ma le informazioni digitali possono essere lunghe milioni di bit e la tecnologia attuale pur essendo diventata capace di leggere un genoma (ordine di grandezza gigabasi), previo spezzettamento per la lettura e riassemblaggio mediante algoritmi, ha un limite nella lunghezza delle molecole di DNA sintetizzabili ex novo (100-200 nucleotidi), cioè non partendo da una copia preesistente.
Vero che questi oligonucleotidi potrebbero essere uniti in una fase successiva a formare molecole più lunghe ma tale approccio non è ad oggi utilizzabile su larga scala. Pertanto, se si volesse usare il DNA come archivio di una informazione digitale, la moltitudine di frammenti separati dovrebbe essere in qualche modo essere leggibile in modo univoco; l'unico modo per farlo sarebbe indicizzare ciascun frammento in modo che l'ordine "di lettura" sia poi facile da ricavare.
Due, quindi, i problemi da affrontare: la scrittura (sintesi di frammenti lunghi o almeno indicizzabili) e la lettura (cioè il sequenziamento).
Sebbene i costi attuali del sequenziamento su larga scala siano una mera frazione dei costi di inizio duemila (l'attuale centinaio di dollari e un operatore necessario contro il miliardo speso per il sequenziamento del primo genoma umano, condotto da un consorzio pubblico e dalla azienda fondata da Craig Venter, per un totale di migliaia di persone coinvolte), il costo ed i tempi sono ancora multipli rispetto al costo di un supporto digitale (vedi sotto)
Il solo CERN ad esempio ha prodotto più di 100 petabyte di dati che devono essere archiviati e resi accessibili non solo per l'immediato ma soprattutto per le future generazioni di ricercatori. Tuttavia i supporti standard per l'archiviazione di dati, come dischi ottici, dischi rigidi e nastri magnetici, hanno una durata variabile ma sempre nell'ordine di anni, se conservati adeguatamente.
 |
| Thanks to Tumisu |
Il DNA, la molecola che codifica le informazioni biologiche, ha il pregio di avere una notevole stabilità (può conservarsi per millenni) e la capacità di immagazzinare informazioni con alto rapporto informazione/densità (può codificare l'equivalente di petabytes di informazioni per grammo).
Lo spunto per l'odierno articolo viene da un lavoro pubblicato su Nature Biotechnology da Lee Organick e colleghi in cui si descrive un metodo per archiviare 200 megabyte di informazioni digitali sotto forma di DNA e di come recuperare tali informazioni senza errori "di lettura".
 |
| Image credit: phys.org |
Fin dagli anni '60, quando dopo la definizione della struttura del DNA da parte di Watson, Crick, Franklin e Wilkins i ricercatori scoprirono il modo con cui l'informazione era codificata (sappiamo oggi che quello era solo il primo livello), gli ingegneri informatici cominciarono a sognare di sfruttare tale sistema per immagazzinare i dati (anche analogici come il testo di un libro) codificandolo sotto forma di DNA (--> Nota 2)
 |
| A (adenosina), T (timidina), C (citidina), G (guanosina) i 4 nucleosidi dell'alfabeto del DNA |
A prima vista, la memorizzazione delle informazioni nel DNA appare semplice. Le informazioni digitali, una serie di 0 e 1 nota come "bit", possono essere facilmente rappresentate come una combinazione di nucleotidi. Ad esempio la sequenza 00-01-10-11 potrebbe essere definita dalla sequenza A-C-G-T, facilmente sintetizzabile.
Se non vedi il video (--> youtube)
Ma le informazioni digitali possono essere lunghe milioni di bit e la tecnologia attuale pur essendo diventata capace di leggere un genoma (ordine di grandezza gigabasi), previo spezzettamento per la lettura e riassemblaggio mediante algoritmi, ha un limite nella lunghezza delle molecole di DNA sintetizzabili ex novo (100-200 nucleotidi), cioè non partendo da una copia preesistente.
Vero che questi oligonucleotidi potrebbero essere uniti in una fase successiva a formare molecole più lunghe ma tale approccio non è ad oggi utilizzabile su larga scala. Pertanto, se si volesse usare il DNA come archivio di una informazione digitale, la moltitudine di frammenti separati dovrebbe essere in qualche modo essere leggibile in modo univoco; l'unico modo per farlo sarebbe indicizzare ciascun frammento in modo che l'ordine "di lettura" sia poi facile da ricavare.
Due, quindi, i problemi da affrontare: la scrittura (sintesi di frammenti lunghi o almeno indicizzabili) e la lettura (cioè il sequenziamento).
Sebbene i costi attuali del sequenziamento su larga scala siano una mera frazione dei costi di inizio duemila (l'attuale centinaio di dollari e un operatore necessario contro il miliardo speso per il sequenziamento del primo genoma umano, condotto da un consorzio pubblico e dalla azienda fondata da Craig Venter, per un totale di migliaia di persone coinvolte), il costo ed i tempi sono ancora multipli rispetto al costo di un supporto digitale (vedi sotto)
A questi problemi pratici si aggiunge un dato non secondario, quello della affidabilità della lettura. Il sequenziamento può non essere sempre univoco, specialmente in corrispondenza di sequenze ricche di C e G, oppure a causa di alterazioni locali; ad esempio la deaminazione spontanea della base C (citosina), fa si che la base modificata venga letta come U (uracile) cioè la base T presente nel RNA causando quindi una mutazione). Per evitare gli errori di lettura della sequenza è necessaria quindi una certa ridondanza della informazione in modo che un eventuale errore venga riconosciuto come tale e neutralizzato. Chiaramente creare ridondanza diminuisce la densità di informazione "utile" a parità di quantità DNA usato.
Ridondanza vuol dire aumentare il numero di molecole di DNA utilizzate e questo rinforza la necessità di sviluppare un sistema di indicizzazione dell'informazione presente (ma dispersa) in una singola provetta.
Nel 2017 (Erlich et al, Science v355) si era riusciti a sviluppare una ridondanza ed un algoritmo di rilevazione errori (quindi di discordanza e di identificazione della informazione corretta) sufficientemente potenti da permettere di archiviare 215 petabyte di informazioni in 1 grammo di DNA. Sebbene il risultato fosse un deciso miglioramento (di almeno 1 ordine di grandezza) rispetto ai precedenti, il metodo aveva un punto debole fondamentale: per estrarre l'informazione di una parte anche minima in essa era necessario "leggere" l'intero archivio.
E' come se volendo leggere la prima terzina del canto X dell'Inferno di Dante, uno dovesse leggere (anche se a velocità supersonica) l'intera Divina Commedia fino a incappare nella frase cercata. O anche leggere un intero disco rigido prima di trovare il file cercato. In un certo senso è quello che capita con i bambini quando si chiede loro di iniziare a recitare la poesia imparata a memoria da una strofa centrale mezzo; per farlo partono dall'inizio.Proprio su questo punto critico ha lavorato il team di Organick riuscendo infine a trovare il modo di archiviare nel DNA e recuperare con successo (e senza "letture inutili") 200 megabyte di dati.
200 MB potranno sembrare risibili per le necessità attuali ma tutto deve essere contestualizzato nell'ambito di una tecnologia emergente. Per mettere tutto in prospettiva, ricordiamoci che il primo disco rigido sul mercato, sviluppato da IBM negli anni '50, era in grado di memorizzare circa 4 megabyte su un dispositivo che pesava più di una tonnellata! Ancora a fine anni 80 il disco rigido fantascientifico a cui uno poteva ambire era di qualche decina di MB)Senza entrare troppo in termini tecnici, per i quali vi rimando all'articolo, ciascun frammento su cui è stata codificata l'informazione digitale è stato legato a "primer" univoci (corte sequenze di DNA, come fossero etichette) in modo che sia possibile, quando necessario, recuperare l'informazione ricercata facendo una sequenza a partire "dall'etichetta" cercata. Nello specifico del lavoro i ricercatori hanno convertito 35 file digitali in un totale di circa 13,5 milioni nucleotidi di DNA suddivisi in frammenti di 150 nucleotidi; il DNA totale è comprensivo della ridondanza interna come sistema di rilevazione degli errori.
 |
| Il principio dell'organizzazione dell'informazione sviluppato nel lavoro di Lee Organick (credit: Organick et al, 2018) |
Il costo attuale per la memorizzazione di un singolo megabyte di dati nel DNA è dell'ordine di un centinaio di dollari, contro meno di 0,0001 $ per anno usando i classici nastri magnetici (in uso nei mega datacenter). Ovviamente il prezzo dell'archiviazione del DNA diminuirà in modo sostanziale al diminuire dei costi di sintesi e di lettura ma questo difficilmente avverrà nella stessa misura per quanto riguarda i tempi dell'analisi. "Scrivere" o "leggere" il DNA è intrinsecamente più lento rispetto al digitale (i millisecondi dei dischi rigidi) per non parlare dell'infrastruttura necessaria, ma questo non è necessariamente un problema se lo scopo è creare archivi di lungo termine e non per la consultazione quotidiana
Con il miglioramento delle tecnologie fondamentali, la molecola che codifica tutte le informazioni biologiche potrebbe diventare un giorno un mezzo robusto, compatto e affidabile per l'archiviazione digitale.
(prossimo articolo sul tema --> "Archiviare le foto nel DNA")
Fonte
- Random access in large-scale DNA data storageRandom access in large-scale DNA data storage
Lee Organick et al, (2018) Nature Biotechnology, 36, pp242–248
***
Nota 1. Da un punto di vista biologico è stato verosimilmente l'RNA il primo depositario di una informazione codificata e trasmissibile e solo in un secondo momento la selezione ha preferito "traslocare" l'informazione sul più stabile DNA. Non a caso molti virus sono basati solo sul RNA.
Nota 2. Rimanendo al primo livello di complessità (trascurando quindi regolazione trascrizionale ed epigenetica), l'informazione genica è codificata da una sequenza di nucleotidi (A, C, G e T) letti come triplette non sovrapposte, delimitate da un segnale di inizio e da uno di termine (tre sono le possibili triplette di "STOP messaggio"). Le triplette possibili sono 64 (43) di cui 61 sono "codificanti", cioè definiscono quale aminoacido dovrà essere inserito durante la sintesi di una data proteina (un gene codifica l'informazione per una proteina). Visto che gli aminoacidi sono 20 e le triplette "utili" 61 ne consegue che alcuni aminoacidi sono codificati da più triplette.
Per quanto riguarda la densità informativa considerate che una cellula umana contiene poco più di 6 picogrammi di DNA (diploide, cioè in doppia copia, ciascuna delle quali originata da un genitore) Di questi in realtà solo l'1,6% è codificante; sebbene un tempo si ipotizzasse che tutto il resto del DNA avesse solo un ruolo strutturale o di "parafulmine" (in cui era più facile che le mutazioni avvenissero lasciando inalterato la parte "utile"), questa idea è stata ampiamente ridimensionata negli ultimi anni grazie alla scoperta del suo ruolo nella regolazione dell'espressione genica (se, quando e dove un dato gene deve essere espresso). Tuttavia per semplificare al massimo il concetto e prendendo per buona la vecchia idea che solo l'1,6% del genoma è utile e dividendolo a metà (genoma aploide, cioè una sola copia di ciascun gene) potrei dire che il nostro "programma" è contenuto in 52 femtogrammi di DNA. Pensate per contrasto quanto "codice" e memoria fisica ci vuole per muovere un robottino capace solo di fare due passi.
Nota 3. Oltre agli errori di lettura bisogna considerare anche le alterazioni a cui il pur stabile DNA può andare incontro con il passare del tempo,
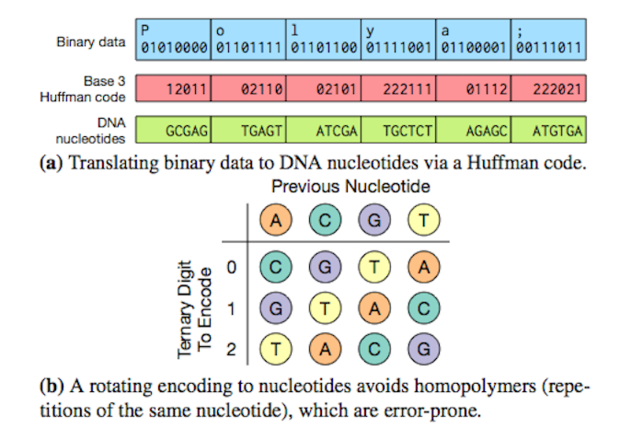
Nota 4. Altro problema è nella non "neutralità" dell'informazione scritta sul DNA rispetto a quella digitale. Per capirci ipotizzando l'associazione C-01 e G-10, non c'è alcun problema in un file 010101010101101010101010. Tuttavia l'analogo CCCCCCGGGGGG (o ogni altra sequenza ricca di CG) pone problemi strutturali a causa della formazione di "forcine per l'appaiamento tra C e G. Stesso discorso per qualunque DNA con lunghe sequenze di nucleotidi identici che pongono un problema in fase di lettura (e anche copiatura/sintesi) noto come "slippage" (scivolamento dell'enzima deputato) che porta alla perdita/aggiunta di nucleotidi e quindi alla modifica dell'informazione
Un modo per evitare la creazione di sequenze ripetute di uno stesso nucleotide (quindi il rischio di "slippage") è quello di usare prima un passaggio intermedio nella codifica da binario a DNA mediante il codice di Huffman e poi una codifica variabile ma predefinita. (credit: University of Washington via computerworld.com)


Nessun commento:
Posta un commento