Di giochi a sfondo scientifico ne ho già scritto in passato e vanno da modalità meramente passive in cui si “regala” la capacità computazionale del proprio computer per la ricerca di segnali extraterresti (vedi SETI@home) a giochi veri e propri l’utente a programmi come Foldit in cui attraverso la risoluzione di puzzle si aiuta a risolvere la struttura 3D della proteina in esame, restringendo i campo per i ricercatori che da questa partiranno per ricavare la struttura definitiva.
Tutti questi sistema sfruttano il potere di calcolo (macchina o umano) fornito spontaneamente dai partecipanti.
Vi rimando a due articoli precedenti sul tema gaming e scienza: "gaming scientifico"; "Con Niche si gioca a fare i biologi evoluzionisti"
Un esempio recente (ed estremo, per numero di persone coinvolte e modalità di analisi) di quello che potremmo definire indirect-science-gaming ha permesso di ricostruire il genoma di un batterio.
Nello specifico il compito è stato svolto da alcuni milioni di giocatori umani che, in simultanea, si sono uniti, in modalità gaming, per risolvere un problema in ambito genomica batterica difficilmente gestibile anche al top delle risorse computazionali disponibili per la ricerca. A costo (quasi) zero e con divertimento dei giocatori; o meglio hanno affrontato il problema senza saperlo visto che per loro si trattava di un puzzle
L’analisi è stata da poco pubblicata su Nature Biotechnology, il che la dice lunga sul livello qualitativo dei risultati ottenuti.
Uno dei problemi principali associati alla elevata capacità produttiva dei moderni sistemi di sequenziamento del DNA è la quantità di dati che vengono prodotti, i quali a loro volta necessitano di potere computazione perché tutte le “stringhe informative” fornite possano essere assemblate nell’ordine corretto. Se, come nel caso del genoma umano (3 miliardi di basi per genoma aploide), si ha già la sequenza di riferimento a cui rapportare la nuova informazione prodotta (ad es. per vedere le differenze individuali) l’analisi è tutto sommato “semplice” in quanto si confronta il dato con il “registro” e si vede se ci sono differenze.
Qualora invece si stia analizzando un genoma “nuovo” come quello di un organismo poco studiato o, come nel caso in esame, un mix di molti genomi microbici diversi, allora il carico computazionale diventa molto elevato.
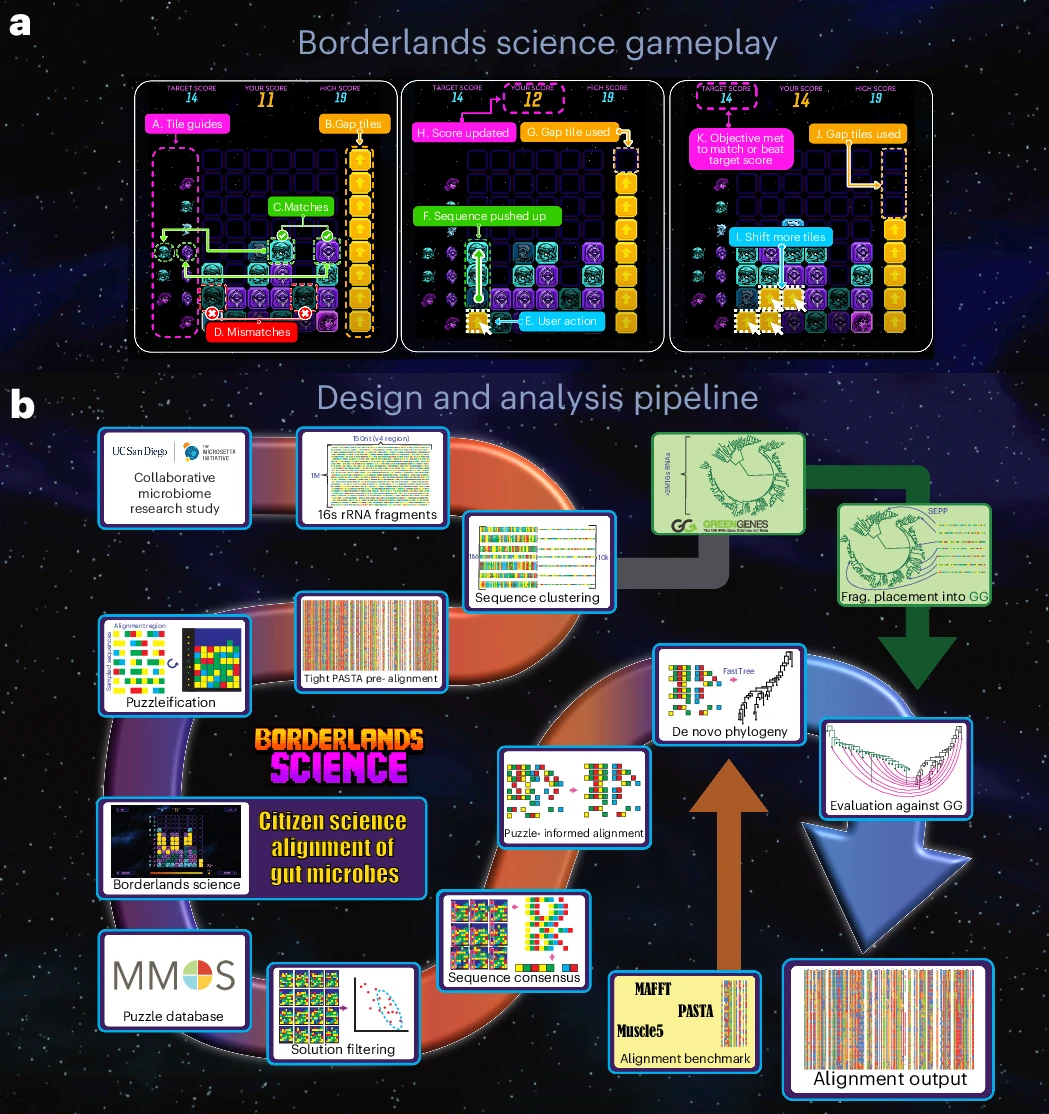
Per facilitare il compito usando "scienziati inconsapevoli", i ricercatori hanno integrato all’interno di un classico sparatutto (Borderlands 3) un puzzle simile ad un tetris, dove era necessario allineare in modo appropriato i mattoncini colorati.
 |
| Il gioco originale in cui è stata inserito il puzzle giocabile via un Arcade credit: Amazon |
Ogni volta che il puzzle veniva risolto, previo accesso ad una arcade inserita nel gioco, i giocatori acquisivano vantaggi nel gioco principale.
 |
| Questo l'arcade che compariva ai giocatori quando (nel gioco) dovevano affrontare un puzzle per guadagnare punti. |
Il corretto allineamento corrispondeva (anche se in modo non esplicito in quanto “mascherato”) all’allineamento di piccole sequenze di DNA ottenute da milioni di campioni ricavati dalla complessa comunità batterica (microbiota) presente nel nostro intestino (a diversi tempi e in diverse condizioni, tipo prima e dopo una dieta). Ognuna delle quattro basi di cui è composto il DNA era rappresentata da un diverso colore del mattoncino e la distribuzione dei mattoncini equivaleva all’allineamento corretto dei tanti frammenti presenti a dare la sequenza complessiva.
 |
| image credit: Roman Sarrazin-Gendron et al / Nature |
In pochi giorni è stato possibile coinvolgere 4,5 milioni di persone che hanno giocato (complessivamente) 135 milioni di volte al puzzle, una potenza di calcolo cumulativa da fare impallidire i supercomputer. Si è così potuto ricostruire il quadro filogenetico rappresentante l’evoluzione di una comunità batterica comunità di batteri che ospitiamo nell’intestino in risposta a cambiamenti di vario tipo.
A differenza di altri giochi, qui si è trattato di utilizzare l’energia computazione nascosta del cervello dei giocatori per risolvere a costo zero compiti che avrebbero richiesto computer molto costosi e tempi lunghi.
Un risultato che dimostra le potenzialità insite nell’affrontare (senza fatica percepita e in modo volontario) un problema complesso unendo milioni di cervelli.
Fonte
- Improving microbial phylogeny with citizen science within a mass-market video game
Roman Sarrazin-Gendron et al, Nature Biotechnology (2024)
***
Per i nerd nostalgici, ecco un libro che ripropone le immagini degli arcade dell'età dell'oro


Nessun commento:
Posta un commento